마스크 포함과 마스크 미포함에 대한 래스터 읽기 속도 비교
본 포스트는 GDAL 유틸리티를 이용하여 래스터 자료를 읽을 때 마스크를 포함한 경우와 마스크를 포함하지 않는 경우의 읽기 속도를 비교 결과를 제시합니다.
GeoTIFF 파일에는 마스크가 라는 것이 있습니다. 마스킹된 부분은 NO DATA 로서 GIS 자료 처리 프로그램에서 특별하게 다루어집니다. 래스터의 격자 값은 Byte, Int32, Float64 등과 같은 실수 값을 갖는데, NO DATA로 된 부분(마스킹된 부분)은 단어 그대로 데이터가 존재하는 않는 것으로 취급합니다. 실제로 정상적인 실수값이 있으나 마스킹 되는 경우가 있는데, 각 격자마다 True or False 값을 갖는 마스킹 레이어를 이용하는 방법과, 특정 값을 NO DATA VALUE 로 지정하고 그 값을 갖는 격자들을 마스킹하는 방법이 있습니다.
Python의 rasterio 패키지를 이용하여 래스터를 읽어들일때 마스킹 여부를 지정할 수 있습니다.
마스킹 없이 읽어들이면 numpy.array 객체가 리턴되고,
마스킹 하여 읽어들이면 numpy.ma.array 객체가 리턴됩니다.
마스킹 데이터와 격자 데이터의 dimension은 서로 동일합니다.
예를들어 1000x1000 크기의 2 차원 GIS 래스터를 읽으면 mask 또한 1000x1000 크기를 가지며,
테이터 타입은 마스킹 여부를 나타내는 True or False 형식입니다. 데이터 해상도와 동일한 마스크를
어떻게든 생성해야 하므로 그에 따른 메모리 사용량과 속도의 저하가 발생할 것이라 생각되며,
그 정도를 측정해 보니 아래와 같았습니다.
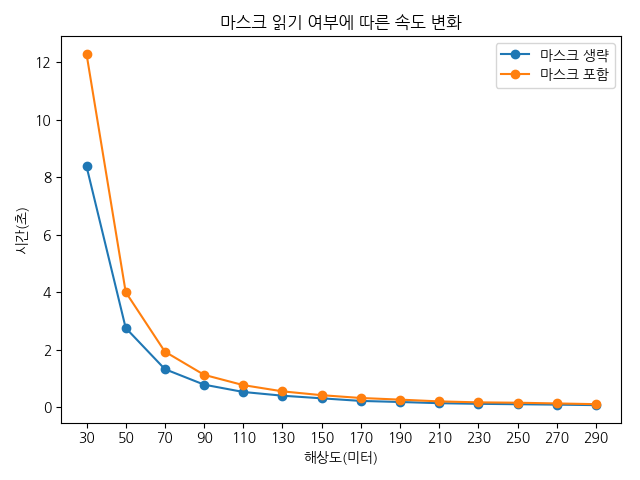
마스크를 읽으면 그렇지 않은 경우 보다 약 1.5배 정도의 속도 향상이 있습니다. 마스크가 필요한 경우가 아니라면 굳이 읽지 않는 것이 나을 것입니다. 그리고 마스크가 있는 래스터는 마스크가 없는 래스터 보다 연산 속도가 더 느립니다. (마스크가 있으면, 마스킹된 격자는 계산하지 않아도 되므로 더 빠를것 같은데, 그 반대입니다. 이에 대한 결과는 향후 제시해 보도록 하겠습니다.)
import rasterio
d = rasterio.open('dem.tiff', masked=True)
rasterio.open 을 이용하여 래스터를 읽을 때 masked=True 로 지정하고 읽을 경우 아래의 GDAL Python 코드와 동일한 속도가 측정되었습니다.
import numpy
import gdal
def read_raster(path, masked=False):
ds = gdal.Open(path)
band = ds.GetRasterBand(1)
data = band.ReadAsArray()
if masked:
mask = band.GetMaskBand().ReadAsArray()
data = numpy.ma.array(data, mask=mask)
return data
d = read_raster('dem.tiff', masked=True)
하지만 특정한 하나의 값으로 마스킹을 수행할 경우 아래와 같은 코드로도 동일한 마스킹 효과를 얻을 수 있으며, 마스크를 읽지 않는 경우와 거의 차이가 없는 속도가 측정되었습니다.
import numpy
import gdal
def read_raster(path, masked=False):
ds = gdal.Open(path)
band = ds.GetRasterBand(1)
data = band.ReadAsArray()
if masked:
mask = data == band.GetNoDataValue()
data = numpy.ma.array(data, mask=mask)
return data
d = read_raster('dem.tiff', masked=True)
Python GDAL을 사용하지 않고 rasterio 코드를 사용하면서도 비슷한 효과를 얻으려면 아래와 같이 작성하면 되겠습니다.
import numpy
import rasterio
r = rasterio.open('dem.tiff')
d = r.read(1)
d = numpy.ma.array(d, mask=(d==r.profile['nodata']))
rasterio 코드와 Python GDAL 코드가 혼재되는 것은 좋아보이지 않습니다. 뭔가 pythonic 하지 않아 보이기도 합니다. 현재로서는 특정한 상황에 맞게 적당한 방법을 사용해야할 것으로 보입니다.
